Pyrrolysine (1/2)
La pyrrolysine, une énigme au sein du vivant
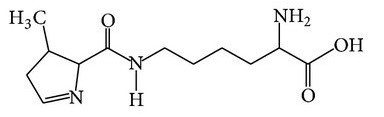
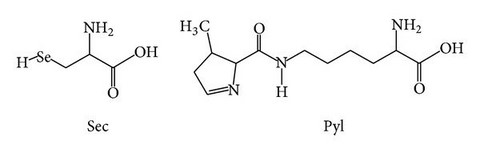
Néanmoins, certains organismes peuvent également utiliser un répertoire élargi d'acides aminés, avec un 21e acide aminé (la sélénocystéine ou Sec), et pour certains autres et rares organismes, la pyrrolysine (ou Pyl). Ces 2 acides aminés particuliers sont représentés sur la figure ci-dessous.
Transcription et traduction de l'information génétique
Ces protéines sont codés par des gènes, dont l'ensemble forme le génome de l'organisme considéré. Ce génome contient l'information de cette identité biologique et permet la transmission de cette identité de génération en génération, sous forme d'acides nucléiques ADN ou acide désoxyribonucléique. L'ADN est formé de 4 formes différentes (les bases A, T, C et G). Pour exprimer ces protéines, un décryptage de l'information contenue dans chaque gène est nécessaire : elle passe tout d'abord par une retranscription du message ADN en un acides nucléiques de nature chimique proche, l'ARN ou acide ribonucléique (transcription en ARNm ou ARN messager, formé à partir des 4 bases A, U, C et G). Puis, ce message va être converti en protéine (traduction) selon des rêgles quasi-universelles à l'ensemble du vivant, le code génétique universel. Cette traduction de l'ARNm s'opère par des structures cellulaires, machineries moléculaires particulières, les ribosomes.
Un code génétique ... quasi universel
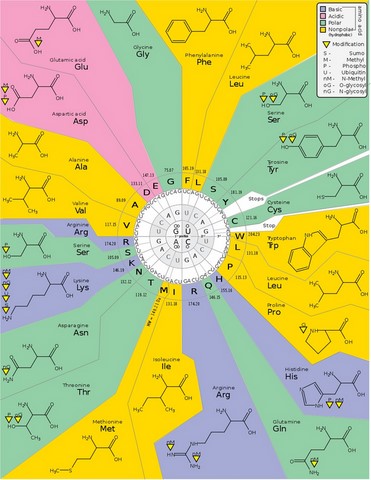
Ce code génétique universel (représenté ci-contre, d'après un illustration de wikipedia) correspond à une relation entre une information de 3 bases (le CODON, sur fond blanc au centre de la figure) traduite en un des 20 acides aminés (représentés ci-contre par les différentes parties colorées). A partir de 4 différentes bases, il existe 64 possibilités de combinaison de 3 bases. Certaines de ces combinaisons correspondent à un même acide aminé (par ex. UCA, UCC,... codant la sérine Ser ou S), et il y a donc une redondance du code génétique. Inversement, une seule combinaison (AUG) code pour la méthionine Met ou M. Ces successions de 3 bases / de codons, sont lues progressivement au niveau du ribosome et permette l'élongation d'une chaine polypetidique, succession linéaire d'acides aminés rajoutés au fur et à mesure de la lecture des codons.
3 de ces 64 combinaisons possèdent une "non-signification", c'est à dire qu'il ne code pas un acide aminé particulier. Au contraire, ils induisent l'arrêt de l'élongation de la chaine polypeptidique et sa libération.
Des adaptateurs assurant la correspondance codon / acide aminé
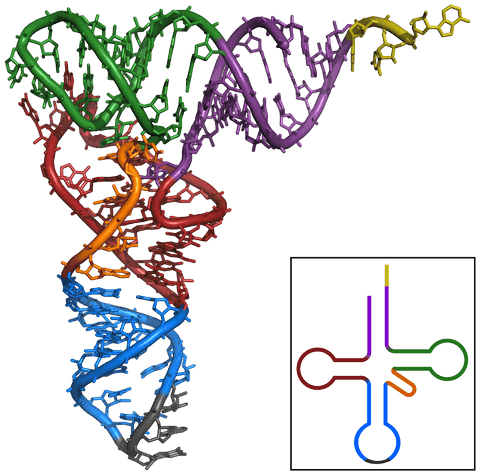
Sur cette figure tirée de Wikipedia, est représenté l'adaptateur assurant la correspondance entre le codon (ARNm) et l'acide aminé (protéine). Il s'agit d'une molécule hybride : elle est d'une part de nature ARN (c'est l'ARNt, ou ARN de transfert), représentée ci-contre en structure tri-dimensionnelle et dans sa forme schématique dite "feuille de trêfle" dans le cadre . Elle présente une zone de 3 bases dite "anticodon", dont la composition est complémentaire et inversée à celle du codon (extrémité basse sur les 2 figures). Elle permet ainsi une reconnaissance spécifique du codon.
D'autre part, cette molécule est associée à une de ces extrémités à un acide aminé exclusif (par la partie représentée en jaune, dite queue CCA), correspondant à celui codé par le codon / par l'anticodon.
La verticalité du code
Un tel code génétique ne peut être efficace que si il y a bien totale adéquation / spécificité totale de l'adaptateur, entre l'anticodon de l'ARNt et la nature de l'acide aminé qu'il porte. Cette verticalité est assurée grâce à des couplages spécifiques entre chaque ARNt et son acide aminé dédié, grâce à des enzymes hautement spécifiques, les AA-ARNt synthetases / aminoacyl-ARNt synthétases / aaRS. Il y a ainsi le plus souvent autant d'aaRS qu'il existe d'acides aminés (habituellement 20) pour assurer sa fixation à la queue CCA de l'ARNt isoaccepteur (des ARNt isoaccepteurs, pour certaines redondances).
Cas de la pyrrolysine
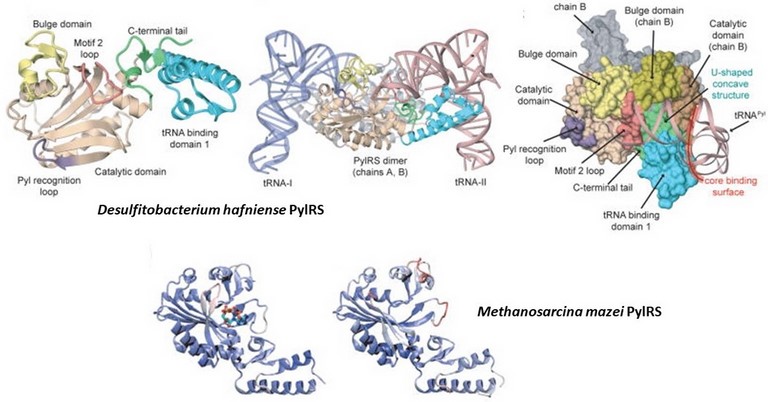
La pyrrolysine n'échappe à ces rêgles universelles. Sur ce lien (page suivante), nous découvrirons l'origine cellulaire de cet acide amnié particulière (comment il est fabriqué par la cellule), la nécessité pour certaines cellules d'avoir ce type d'acide aminé, et quelle est sa distribution dans le vivant. L'orthogonalité de ce code génétique permettant d'encoder la pyrrolysine est garantie par une enzyme, la Pyl-tRNA Synthetase (PylRS) : elle possède une reconnaissance "spécifique" de la pyrrolysine et une reconnaissance "spécifique" de l'anticodon correspondant au codon unique codant la pyrrolysine. Beaucoup de guillements... car ce système d'encodage est en fait très particulier, voir énigmatique. Cette enzyme PylRS, telle qu'elle est rencontrée chez la bactérie Desulfitobacterium hafniense et l'archée méthanogène Methanosarcina mazei, est représentée ci dessus, selon les travaux du groupe de Dieter Söll et de P. O'Donoghue (ici, figures 1 et 4 de l'article de Nozawa et al., 2009 accessible ici).