Technologies développées
des simulateurs d'environnement du microbiote intestinal humain
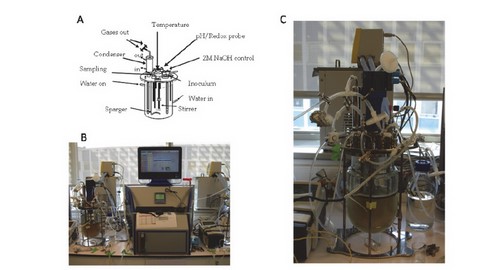
Simuler le plus fidèlement possible le fonctionnement du microbiote intestinal humain en "tube à essai", tel est le défi pour pouvoir l'étudier plus facilement, à la fois au niveau scientifique et au niveau éthique. A cette fin, outre l'ensemble des outils développés par la plateforme in vitro d'outils digestifs, nous avons développé des fermentations continues dans des conditions environnementales (pH, T°, nutriments disponibles,...) retrouvées dans différentes régions anatomiques du colon humain, ensemencées à partir de microbes récoltés de selles humaines.
des méthodes originales de culture des microbes anaérobies EOS

La culture, et mieux encore, la culture de microorganismes isolés ou en consortium limités et déterminés, permettent l'étude fine de la biologie des microorganismes vivant dans nos intestins. Cependant, ces techniques doivent être aplliqués à des organismes vivant en absence d'oxygène (anaérobie stricts), et pire encore, extrèmement sensibles à l'oxygène (EOS, extremely sensitive organisms). Pour cette raison, des techniques particulières de microbiologie anaérobie doivent être employées, nécessitant un appareillage et une technicité particulière.
Grâce à l'expertise de Bernard OLLIVIER (DR IRD, MIO-MEB, Marseille) et de Marie-Laure-Fardeau, Wajdi Ben Hania et le groupe a pu mettre en évidence des propriétés inattendues des archées méthanogènes du microbiote intestinal humain et considérer ainsi leur culture axénique (culture pure d'un membre isolé) et leur production en quantité suffisante à leur étude: ce sont des préambules nécessaires à un développement de ces organismes comme probiotiques et permet de sélectionner les souches les plus prometteuses.
des outils de détection des composants du microbiote intestinal
Le microbiote intestinal humain est formé de plus de 100 milliards de cellules, en majorité des bactéries (env. 98%) et des archées (env. 2%). Les techniques microbiologiques classiques, essentielles pour pouvoir étudier la biologie de ces organismes commensaux et leurs relations entre eux, est malheureusement trop lourde à employer pour des études systématiques, et souvent également inopérantes pour de nombreux microorganismes (voir le chapitre ci-dessous). Pour cette raison, le groupe a développé des approches de biologie moléculaire, par lesquelles l'identification et la quatification des microorganismes sont effectuées à partir de la présence de leur ADN. Outre les approches devenues classiques de séquençage de nouvelle génération (NGS) du marqueur moléculaire microbien 16 S, le groupe a notamment développé des amorces qPCR pour les archées méthanogènes (PCR quantitative en temps réel) et une biopuce phylogénétique, exploratoire: la HuG-Chip ou Human Gut Chip.
Pour de plus amples informations sur la HuG-Chip, voir la rubrique "Travaux",
Tottey W., Denonfoux J., Jaziri F., Parisot N., Missaoui M., Hill D., Borrel G., Peyretaillade E., Alric M., Harris H.M.B., Jeffery I.B., Claesson M.J., O’Toole P.W., Peyret P., Brugère J.-F. (2013).
The human Gut Chip “HuGChip”, an explorative phylogenetic microarray for determining gut microbiome diversity at Family level.
PlosONE, 8(5): e62544.