Les Methanomassiliicoccales intestinales
Cette page est encore en évolution.... n'hésitez pas à revenir bientôt (11/02/2018)
Les Methanomassiliicoccales, qu'est-ce ?
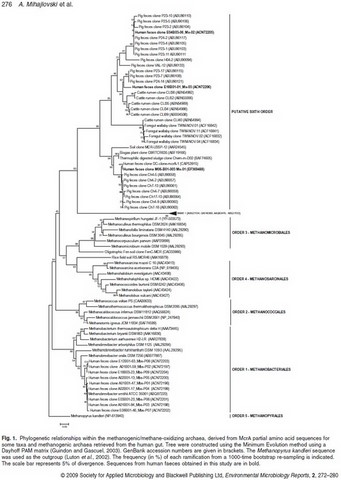
- aucun autre méthanogène dans cette branche évolutive à ce moment là, rendant cette hypothèse incompatible avec les connaissances de l'origine de la méthanogenèse et de son évolution dans le vivant. Comme le montre la figure ci-contre, toutes les archées méthanogènes (en jaune) étaient toutes issues d'une branche centrale, rendant peu probable que les Methanomassiliicoccales soient des organismes méthanogènes (en jaune à gauche, proches des Thermoplasmatales).
- aucun argument biochimique, métabolique et moléculaire d'une méthanogenèse
- pas d'organismes isolés ou même cultivés permettant de tester ces hypothèses métaboliques.
2008 et 2010: Des méthanogènes inconnus dans le microbiote humain
L' étude publiée en 2017 par Guillaume Borrel dans le laboratoire de P. W. O'Toole (UCC-APC, Cork; Ireland) en collaboration avec notre groupe confirme la présence chez l'Homme de ces 5 espèces de Methanomassiliicoccales. Elle est le fruit d'une analyse approfondie de bases de données de séquences, de la bibliographie et surtout d'une centaine de métagénomes (étude ELDERMET). Il reste néanmoins possible que d'autres espèces soient présentes. Il est surtout maintenant avéré que ces espèces n'ont pas le même potentiel génétique et métabolique, et qu'une grande variabilité génomique semble exister au sein d'une même espèce (Borrel et al, 2017).
Un isolat, des génomes : premiers éléments de l'originalité de ces organismes
Une première espèce de Methanomassiliicoccale a été isolée en 2012 par les efforts combinés du laboratoire de Didier Raoult et de celui de Bernard Ollivier (Dridi et al, 2012). Le nom de cette espèce, attribué par les découvreurs est Methanomasiliicoccus luminyensis. Il induit de fait automatiquement le nom donné à la famille (rang taxonomique supérieur) et à l'ordre (rang taxonomique encore au dessus) si l'espèce ne peut être rattachée à un rang taxonomique déjà décrit : ainsi est né la famille des Methanomassiliicoccaceae et l'ordre des Methanomassiliicoccales. Le groupe de Didier Raoult a également déterminé la quasi-intégralité du génome de cet isolat (Aurore Gorlas et al, 2012), qui est un génome de grande taille (env. 2,7 Mpb). Son annotation a été réalisée de manière automatique / non-experte par GenBank (détermination des gènes, des fonctions supposées pour lesquels ils codent, ...). Un mois après que ce génome ait été annoncé, nous publions également le génome d'une Methanomassiliicoccale d'un genre différent (Ca. Methanomethylophilus alvus, Borrel et al, 2012) puis d'une autre espèce, mais probablement à rattacher au même genre (Ca. Methanomassiliicoccus intestinalis, Borrel et al, 2013) et en collaboration avec Simonetta Gribaldo (Institut Pasteur Paris) apportions les preuves défintives, sur des bases phylogénomiques, que les Methanomassiliicoccales formaient bien un ordre méthanogène à part entière sur une branche inattendue du vivant (Borrel et al, 2013).
Une activité méthanogène très originale
Dès les premiers éléments de connaissance des Methanomassiliicoccales connus, leur méthanogenèse a interrogé :
- l'isolat M. luminyensis (2012) montre une méthanogenèse originale réalisée à partir de méthanol (comme pour les méthanogènes méthylotrophes) mais nécessitant de l'H2 comme donneur d'électron (Dridi et al, 2012). Cette exception n'est pas unique, d'autant moins pour un habitant du microbiote intestinal, car ce même type de méthanogenèse est réalisé par Methanosphaera stadtmanae, une méthanobacteriale retrouvée dans les microbiotes digestifs notamment (voir ici).
- L'analyse du génome de Ca. Methanomethylophilus alvus (Borrel et al, 2012), puis d'autres ont rapidement établi que les gènes pour une méthanogenèse dite "hydrogénotrophe" étaient totalement absents (Borrel et al, 2013); De plus, une première analyse comparative des génomes disponibles (3 en 2013-2014) a montré l'absence de certains éléments permettant les transferts d'électrons / de protons et la récupération d'énergie une fois la molécule de méthane fabriquée (Borrel et al, 2014). Différentes hypothèses ont donc été émises (Borrel et al, 2014 ; Lang et al, AEM, 2015). En particulier, les travaux du groupe de Uwe Deppenmeier en Allemagne permettent de proposer un nouveau mécanisme, étayé par des données expérimentales pour Methanomassiliicoccus luminyensis (Kröninger et al, FEBS Journal, 2016) .
- Dès le génome de Ca. Methanomethylophilus alvus connu (Borrel et al, 2012), nous avons émis l'hypothèse que cette espèce pouvait utiliser d'autres composés simples méthylés que le méthanol. En particulier, les gènes permettant l'utilisation des méthylamines (mono-, di- et triméthylamine, soit MMA, DMA et TMA) ou du diméthyl sulfide (DMS) sont présents, comme c'est le cas chez les Methanosarcinales de la famille des Methanosarcinaceae, celles qui réalisent une méthanogenèse méthylotrophe "pure".
- Par ailleurs, le métabolisme des méthylamines en méthane nécessite des enzymes très originales : ce sont des méthyltransférases (Mt) présentant dans leur site actif un acide aminé rare, la pyrrolysine ou Pyl. L'acquisition de cet acide aminé se fait par un code génétique modifié, faisant de la pyrrolysine le 22e acide aminé protéinogène, alors que les êtres vivants possèdent habituellement une base de 20 acides aminés protéinogènes, codés par un code génétique quasi universel. Le cas des Methanomassiliicoccales que nous avons pu étudier jusqu'à maintenant est très original (Borrel et al, 2014b), expliquant que nous y consacrions une partie de nos travaux.
- Nous avons apporté les preuves expérimentales que cette méthanogenèse pouvait se réaliser à partir des méthylamines à partir de la souche isolée initialement (Brugère et al, 2014), ainsi que pour un membre de l'espèce Methanomethylophilus alvus (Borrel et al, 2017). Cependant, certaines Methanomassiliicoccales n'utilisent pas toutes les méthylamines (voir par exemple chez le termite, une souche n'utilisant pas la di- et triméthylamine, Lang et al, 2014). Ceci est également apparemment vrai dans le tube digestif de l'homme: certaines Methanomassiliicoccales n'ont pas les capacités génétiques d'utiliser la di- et la triméthylamine, ou même l'intégralité des méthylamines simples Borrel et al, 2017). Par ailleurs, ces dernières n'ont pas de système pyrrolysine.
Les Methanomassiliicoccales, Deux lignages pour deux types d'environnement?
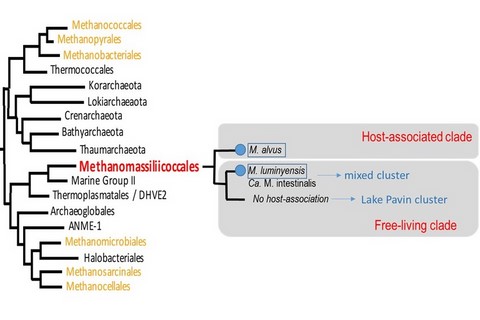
D'une manière assez surprenante, les Methanomassiliicoccales semblent pouvoir être divisées en 2 grands lignages. L'un est constitué apparement d'espèces retrouvées que dans des environnements digestifs (insectes, ruminants, homme, ...) alors qu'un second lignage est principalement composé d'espèces retrouvées dans des environnements très divers tels que les sédiments de lac d'eau douce ou de fonds marins, dans les tourbières, etc... (Borrel et al, 2013 ; confirmé par Söllinger et al, 2016 ou Borrel et al, 2017). Le premier lignage composé est dit "associé à un hôte", ou "Host-associated clade". Le second est nommé en opposition "Free-living clade", groupe ne semblant pas vivre avec un hôte. Il y a cependant 2 exceptions actuellement connues de membres du clade "free-living" vivant chez un hôte:
- Methanomassiliicoccus luminyensis appartient à ce clade, alors qu'il a été isolé de selles humaines, et n'a pas été retrouvé pour l'instant dans d'autres environnements. Cependant, il est difficile d'affirmer qu'il soit présent fréquemment chez l'homme: en dehors de son isolement, il n'a pas été retrouvé de manière sûre chez l'homme, les outils employés pouvant permettre de confondre avec un deuxième membre de ce clade que l'on sait présent chez l'homme.
- Ce deuxième membre est l'espèce candidate Ca. Methanomassiliicoccus intestinalis. D'une manière surprenante, il semble associé à l'homme âgé en état de santé précaire où il semble surpasser / remplacer les membres du clade "Host-associated". Quelles en sont les conséquences et les origines ? En tout état de cause, cette espèce interroge sur un possible rôle de pathobionte pour cette espèce archéenne.